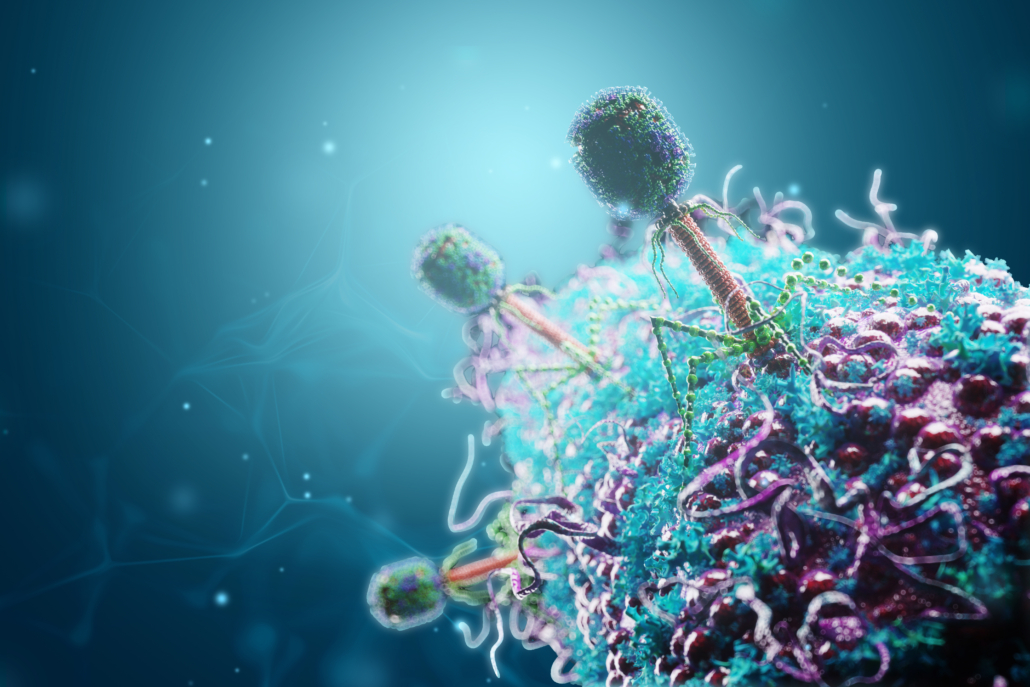
Mit Phagen gegen multiresistente Erreger
Ein Forschungsteam aus Tübingen hat eine Methode entwickelt, mit dem sich Bakteriophagen identifizieren lassen, die spezifisch gegen gefährliche Krankheitserreger gerichtet sind. Dieser Ansatz könnte helfen, Antibiotika zu ersetzen.
Die Therapie von gefährlichen Infektionen mit Hilfe spezifischer Bakterienviren (Phagen) kommt in Deutschland noch nicht standardmäßig zum Einsatz. Dabei könnte sie eine Alternative zur Behandlung mit Antibiotika sein. Zu solchen Phagentherapien könnte eine im Fachjournal Cell Reports vorstellte Methode zur Identifizierung neuer viraler Rezeptorbindeproteine beitragen, mit deren Hilfe Phagen Staphylococcus aureus binden und infizieren können.
Multiresistente Krankheitserreger werden zunehmend zur Bedrohung für die globale Gesundheit. Während an der Kriegsfront in der Ukraine immer gefährlichere Stämme von Klebsiella pneumoniae gefunden werden, bleiben Methicillin-resistente S. aureus-Erreger (MRSA) ein Problem vor allem in Krankenhäusern, wo sie schwere Entzündungen und Sepsis auslösen können. In diesen Erregern entwickeln sich Resistenzen gegenüber einer Reihe von Antibiotika sehr schnell aus bereits vorliegenden Mutationen, wie eine andere aktuelle Studie zeigte. „Antibiotika sind eine unserer wichtigsten Waffen gegen Infektionen, aber wir sehen immer häufiger, dass sie entweder nicht mehr anschlagen oder zu viele Nebenwirkungen verursachen“, so Studienleiter Andreas Peschel von der Universität Tübingen. „Phagen hingegen sind hochspezifisch und können gezielt einzelne Erreger eliminieren, ohne das restliche gesundheitsfördernde Mikrobiom der Patienten zu stören.“
Am Exzellenzcluster „Kontrolle von Mikroorganismen zur Bekämpfung von Infektionen“ (CMFI) der Universität Tübingen forscht er mit seinem Team zu den Interaktionen von Bakterien mit anderen Organismen und wie sie sich für Therapien anwenden lassen. Um passende Phagen für eine Therapie zu identifizieren, entwickelten die Autoren ein Tool namens PhARIS (Phage Aureus RBP Identification System), mit dem sich Rezeptorbindeproteine (RBPs) identifizieren lassen, die Phagen für die Erkennung und Infektion ihrer Wirtsbakterien benötigen. Die Methode analysiert diverse Phagengenome und erkennt anhand spezifischer Bindeproteine, ob und wie effektiv ein Phage eine bestimmte S. aureus-Variante infizieren kann. Auf diese Weise ließen sich 335 solcher RBPs identifizieren, die gegen Wandteichonsäuren (WTA) des Wirtsbakteriums gerichtet sind. Die meisten Phagen kodierten davon zwei verschiedene Varianten. Weitere Analysen ergaben acht verschiedene RBP-Cluster, die experimentell auf ihre Bindefähigkeit hin untersucht wurden. Basierend darauf gelang es dem Team, ein Tool zur Vorhersage der Bindefähigkeit aus der Genomsequenz zu entwickeln.
Die so identifizierten Bakteriophagen könnten bei einer Phagentherapie zukünftig gezielt bestimmte krankheitserregende Bakterienvarianten infizieren und zerstören. Sobald alle Bakterien abgetötet sind, können sich auch die Phagen aufgrund ihrer Spezifität nicht weiter vermehren. Der Ansatz könnte somit eine Ergänzung zu einer möglichen Therapie mit dem „Phagenlysin“ HY-133 darstellen – einem Designer-Phagenprotein der HYpharm GmbH, das hochspezifisch MRSA-Stämme auflöst und gerade in ersten klinischen Studien getestet wird.
Das PhARIS-System soll nun für weitere Erreger weiterentwickelt werden und so zu einem Standardwerkzeug für Labore werden, um Phagen schnell und effektiv als therapeutische Alternative zu Antibiotika einsetzen zu können.

 Smartbax GmbH
Smartbax GmbH AMR Action Fund
AMR Action Fund LimmaTech Biologics AG
LimmaTech Biologics AG