Neuer Ansatz enthüllt RNA-Funktion in Immunzellen
Mit einem innovativen Analyseverfahren haben Marburger Forscher ein bislang verborgenes Netzwerk aus nicht-kodierenden RNAs und Proteinen entschlüsselt und damit die gezielte Steuerung von Entzündungsprozessen in Immunzellen sichtbar gemacht.
Sie galten als nutzloser Rest des Genoms – lange nicht-kodierende RNAs (lncRNAs). Doch in den vergangenen Jahren mehrten sich Hinweise, dass sie wichtige Funktionen übernehmen – etwa in der Genregulation. Neue Untersuchungen eines Forschungsteams um Professor Dr. Leon Schulte von der Philipps-Universität Marburg zeigen nun: Sie spielen eine zentrale Rolle in der Immunabwehr gegen bakterielle Infektionen. Mit Hilfe eines neu entwickelten Analyseverfahrens konnte das Team erstmals umfassend darstellen, wie lncRNAs mit Proteinen in Makrophagen zusammenwirken. In Nature Communications veröffentlichten die Wissenschaftler nun einen detaillierten Atlas dieser bislang unbekannten molekularen Netzwerke.
„Wir konnten zeigen, dass viele dieser RNAs direkt in Immunprozesse eingreifen und dabei helfen, Entzündungsreaktionen gezielt zu steuern“, erklärt Schulte. Seine Arbeitsgruppe untersucht seit Jahren, wie nicht-kodierende RNAs die Immunantwort regulieren. Mit ihrem multidimensionalen Ansatz gelang es den Forschern erstmals, die funktionellen Netzwerke aus nicht-kodierenden RNA und Proteinen in Makrophagen systematisch zu kartieren.
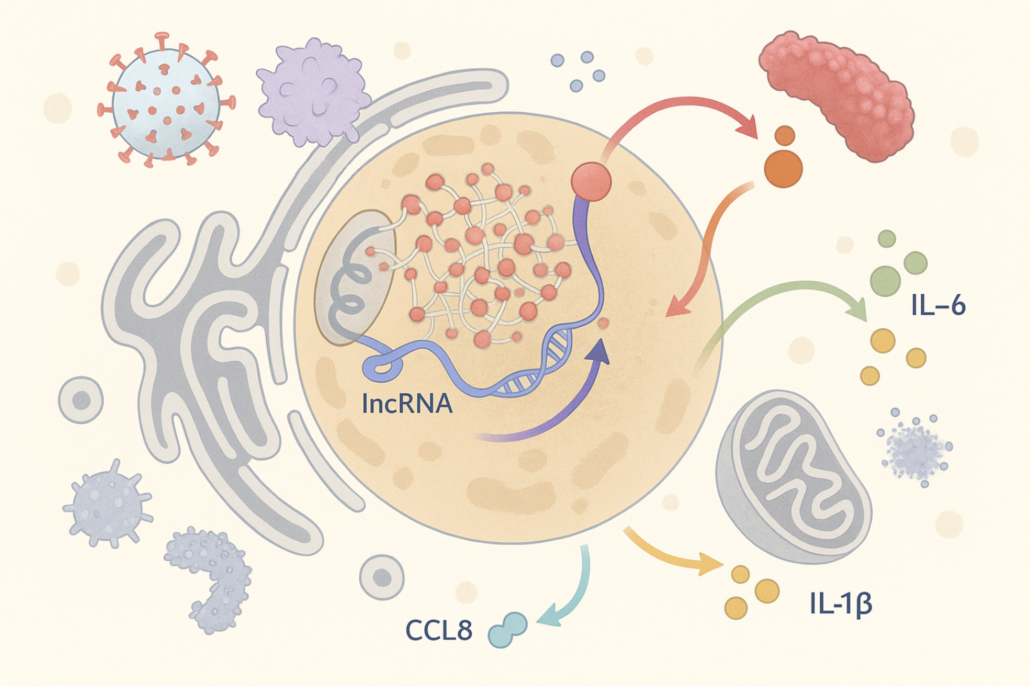
Besonders auffällig ist das lncRNA-Molekül ROCKI: Es blockiert gezielt einen zellulären „Aus-Schalter“, der normalerweise Entzündungsreaktionen dämpft, – mit dem Ergebnis, dass die Immunantwort verstärkt wird. Konkret verhindert ROCKI die Fehlaktivierung des Gens GATA2, das eigentlich nur in Vorläuferzellen aktiv ist. In reifen Makrophagen hemmt GATA2 die normale Entzündungsfunktion – seine Blockade durch ROCKI schützt daher die volle Funktionsfähigkeit dieser Immunzellen. Dadurch beeinflusst ROCKI auch die Produktion wichtiger Entzündungsproteine wie IL-6, IL-1β und CCL8. Daneben identifizierte das Forschungsteam vier weitere, bislang kaum untersuchte lncRNAs, die zentrale Signalwege der angeborenen Immunabwehr beeinflussen. „Unsere Ergebnisse zeigen, dass lange nichtkodierende RNAs ein bislang unterschätztes Steuerungselement in der menschlichen Immunabwehr darstellen“, so Schulte
Möglich wurde dieser Durchbruch durch ein neu entwickeltes Hochdurchsatzverfahren namens GRADR (Genome-wide RNA–protein interaction Analysis with Direct RNA labeling). Die Methode kombiniert RNA-Sequenzierung, CRISPR-basierte Genmanipulation und quantitative Proteomanalysen, um gezielt nach Wechselwirkungen zwischen lncRNAs und Proteinen in Immunzellen zu suchen. Im Kern basiert GRADR auf der Gradienten-Co-Sedimentation: RNAs und Proteine, die in denselben Fraktionen auftreten, gelten als potentielle Interaktionspartner. Das Verfahren erlaubt es erstmals, mögliche Bindungspartner einer Vielzahl nicht-kodierender RNAs in nur einem Experiment systematisch zu identifizieren. Damit liefert GRADR eine skalierbare, experimentell gestützte Grundlage zur Aufklärung bislang unbekannter RNA-Protein-Netzwerke im Immunsystem.
Auf der frei zugänglichen Webplattform SMyLR (Systematic Map of lncRNA Regulation) stehen die Daten zur Verfügung. Forscher können gezielt nach Immun-RNAs suchen, deren regulatorische Mechanismen nachvollziehen und potentielle Proteinpartner identifizieren. Langfristig sollen die Erkenntnisse dabei helfen, neue Diagnoseverfahren und Therapieansätze für entzündliche Erkrankungen wie Sepsis, Lungenentzündung oder chronisch-entzündliche Autoimmunerkrankungen zu entwickeln. Die neu entdeckten RNA-Protein-Netzwerke bieten dafür erstmals konkrete Angriffspunkte.
Parallel zeigt das starke Interesse von Unternehmen wie Haya Therapeutics, das mit einer frischen Finanzierungsrunde von 65 Mio. US-Dollar seine erste lncRNA-basierte Therapie gegen Herzinsuffizienz vorantreibt, dass das regulatorische Genom zunehmend als vielversprechender Ansatz für innovative Therapien erkannt wird.

 Haidong Yu
Haidong Yu unknownuserpanama auf Pixabay
unknownuserpanama auf Pixabay Kuros Biosciences AG
Kuros Biosciences AG