Über Datenknotenpunkte zu unerkannten Verbindungen
Die Vielzahl von Verknüpfungen und Zusammenhängen der molekularen Ebenen von Krankheiten haben Forscher aus München und Kanada in ein Wissensdatennetz gepackt, das auch bisher unbekannten Beziehungen nachspürt.
Forscher von Helmholtz München und dem Mila-Quebec Artificial Intelligence Institute haben ein KI-Verfahren entwickelt, das verborgene Zusammenhänge in großen biomedizinischen Datennetzen aufspüren soll. Das System namens BioPathNet wurde jetzt in Nature Biomedical Engineering vorgestellt.
Datenknoten mit Knowledge-Graph
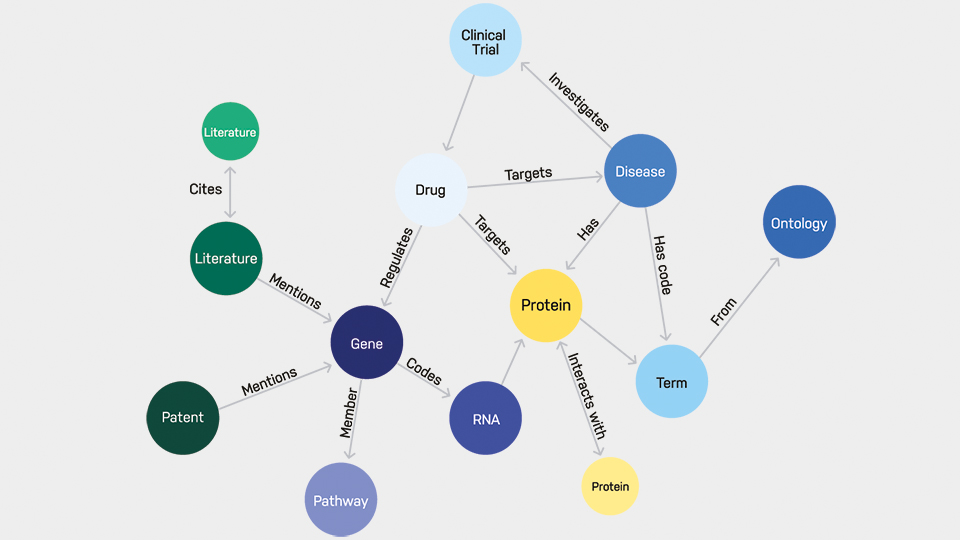
Im Zentrum stehen sogenannte biomedizinische Wissensgraphen. Darunter kann man sich ein mehrdimensionales Gitterknotennetz vorstellen wie in einem Kristall, wo von jedem Knotenpunkt nachvollziehbare Verknüpfungen zu weiteren Knotenpunkten ausgehen. An den Knotenpunkten können ganz unterschiedliche Wissenssammlungen zusammengeführt werden. Mit dieser Technologie hatte beispielsweise der Graph-Experte Neo4J die Panama-Papers durchforstet und die kaum zu entwirrenden Verbindungen von Personen und Unternehmen sortieren können. Im Fall von BioPathNet verknüpfen sich an den Graphknoten Gene, Proteine, Krankheiten, Wirkstoffe und biologische Prozesse miteinander und bilden so vorhandenes Wissen strukturiert ab. Allerdings sind diese Netze unvollständig – viele potenzielle Zusammenhänge sind bislang nicht explizit erfasst.
BioPathNet setzt genau hier an. Anders als herkömmliche Ansätze analysiert das Modell nicht nur einzelne Knoten oder direkte Verbindungen, sondern ganze Beziehungsketten – etwa vom Gen über einen Signalweg zu einer Erkrankung und weiter zu einem möglichen therapeutischen Ansatz. Aus tausenden solcher Muster lernt das System, welche neuen, biologisch plausiblen Verknüpfungen wahrscheinlich sind. Ein zentrales Merkmal ist dabei die Nachvollziehbarkeit: Für jede Vorhersage lässt sich rekonstruieren, über welche Pfade im Graphen sie zustande kam.
Neue Positionen für bekannte Wirkstoffe
In Tests nutzte das Team um Annalisa Marsico das Verfahren für unterschiedliche Fragestellungen von der Vorhersage von Genfunktionen über Krankheitszusammenhänge bis hin zu potenziellen Angriffspunkten in der Krebstherapie und neuen Einsatzmöglichkeiten vorhandener Medikamente. Bei komplexen Erkrankungen wie Leukämien, Magenkarzinom und Alzheimer identifizierte BioPathNet sowohl etablierte Therapien als auch Substanzen, die sich bereits in klinischer Prüfung befinden.
Die Forscher verstehen BioPathNet ausdrücklich nicht als automatisches Entscheidungssystem, sondern als Hypothesengenerator. Jede vorgeschlagene Verbindung müsse experimentell oder klinisch überprüft werden, zudem hänge die Aussagekraft von der Qualität der zugrunde liegenden Daten ab. Perspektivisch sehen sie das Verfahren als Baustein für umfassende, flexibel anpassbare Grundmodelle biomedizinischer Wissensgraphen – etwa für die Wirkstoffsuche oder die Analyse komplexer Krankheitsmechanismen.
Entstanden ist BioPathNet aus einer interdisziplinären Zusammenarbeit zwischen Computational Biology und Machine Learning. Methodisch basiert das System auf dem Graph-Neural-Network-Framework NBFNet. Es ist als Open-Source-Lösung verfügbar.
Literatur:
Hu et al., 2026: Enhancing link prediction in biomedical knowledge graphs with BioPathNet. Nature Biomedical Engineering. DOI: 10.1038/s41551-025-01598-z

 PK Fotografie
PK Fotografie Care for Rare Stiftung
Care for Rare Stiftung Helmholtz Zentrum München
Helmholtz Zentrum München