Moscot entschlüsselt Zelldynamik
Mit dem bahnbrechenden Moscot-Ansatz lassen sich Bewegungen und Veränderungen von Millionen von Zellen gleichzeitig in ihrer räumlich-zeitlichen Entwicklung nachvollziehen.
Moderne Einzelzellanalysen liefern Unmengen von Daten, deren Interpretation jedoch schwierig ist. Die komplexen Wechselwirklungen unzähliger Zellen in Raum und Zeit während der Organentwicklung oder bei Krankheitsprozessen etwa lassen sich mit bisherigen Methoden nur schwer nachvollziehen, da sie lediglich eine Momentaufnahme liefern können. Ein internationales Forschungsteam hat nun im renommierten Fachjournal Nature ein neues Software-Framework vorgestellt, das enorme Datenmengen mit Hilfe komplexer Algorithmen analysiert und gleichzeitig einen intuitiven Zugang für Wissenschaftler bietet.
Die innovative Methodik namens Moscot (Multi-Omics Single-Cell Optimal Transport) ermögliche nicht nur, die Dynamik der Zellentwicklung in bisher unerreichter Detailtiefe zu erfassen. Sie erlaube auch präzise Vorhersagen über die Entwicklung von Krankheiten – wie Letztautor Fabian Theis verdeutlicht: „Moscot verändert die Art und Weise, wie wir biologische Daten verstehen und nutzen.“ Der Direktor des Institute of Computational Biology bei Helmholtz Munich und der TU München ist Experte für die mathematische Modellierung biologischer Prozesse. Im Jahr 2023 wurde er mit dem Leibniz-Preis ausgezeichnet. Bei der Entwicklung von Moscot arbeitete er mit einem internationalen Team aus Mathematikern und Biologen sowie KI-Experten von Apple und Google DeepMind zusammen.
Alte Theorie neu gedacht
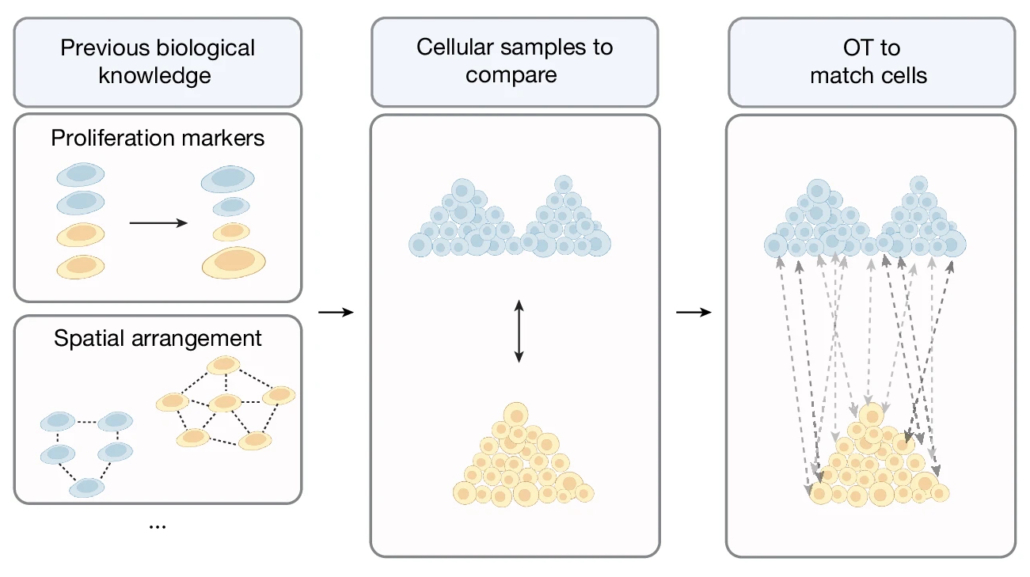
Die Moscot-Analyse beruht auf einem Optimierungsproblem aus dem 18. Jahrhundert: Die Theorie des optimalen Transports beschreibt, wie sich Dinge möglichst effizient von einem zum anderen Ort bewegen lassen. Übertragen auf Zellpopulationen hilft sie zu verstehen, wie Zellen sich bewegen, verändern oder von einem in einen anderen Zustand übergehen. Mit Hilfe künstlicher Intelligenz lässt sich die Theorie nun auf riesige biomedizinische Datensätze anwenden. Moscot erlaubt eine multimodale Zuordnung verschiedener experimenteller Daten von unzähligen Einzelzellen im Gewebe. Dabei erfasst die Analyse simultan den molekularen Zustand der Zellen, um deren Entwicklung in Raum und Zeit zu beschreiben.
Die in Nature vorgestellte Arbeit zeigt auch die Anwendungsmöglichkeiten von Moscot. Das Team nutzte verschiedene experimentelle Datensätze und konnte damit die Entwicklung hormonproduzierender Zellen der Bauchspeicheldrüse kartieren. Durch zeitliche Verknüpfung von Millionen Zellen ließen sich Veränderungen in der Genexpression mit Zellentscheidungen in Beziehung setzen. Auf dieser Grundlage könnten nun die zugrundeliegenden Mechanismen von Diabetes besser verstanden und so zielgerichtete Therapien entwickelt werden.
Omics-Datenanalyse und virtuelle Zellen
Die rasante Entwicklung in der Datenanalyse unterstreicht die Bedeutung künstlicher Intelligenz für biomedizinische Anwendungen. Bereits in einer anderen aktuellen Arbeit in Nature Machine Intelligence hatte ein Team um Fabian Theis aufgezeigt, wie maschinelles Lernen dazu beiträgt, riesige Datensätze neu zu interpretieren und aussagekräftige Informationen aus den Mustern zu erkennen. Besonders für sogenannte unbeschriftete Daten, die in verschiedenen Omics-Ansätzen entstehen, eignet sich selbstüberwachtes Lernen am besten, wie die Autoren an Genexpressionsdaten von über 20 Millionen Zellen demonstrierten.
Eine Kombination der Datenrepräsentation durch maschinelles Lernen mit der Modellierung dynamischer Prozesse durch Moscot könnte die Analyse von Zellveränderungen weiter verbessern. Auch für virtuelle Zellen – umfassende Computermodelle, die eine Vielfalt von Zellen in verschiedenen Datensätzen abbilden – sind die aktuellen Arbeiten von Theis‘ Arbeitsgruppe von enormer Bedeutung. Sie zeigen, wie sich solche Modelle effizienter trainieren lassen, und bringen deren Realisierung entscheidend voran.

 Flinn.ai
Flinn.ai geralt/pixabay.com
geralt/pixabay.com Chormail – Freepik.com
Chormail – Freepik.com