3D-Kartierung von Tumoren
Durch die Kombination räumlicher Einzelzelltechnologien ist es Berliner Wissenschaftlern gelungen, die Nachbarschaft von Tumorzellen dreidimensional zu kartieren – mit neuen Perspektiven für die zielgerichtete Krebstherapie.
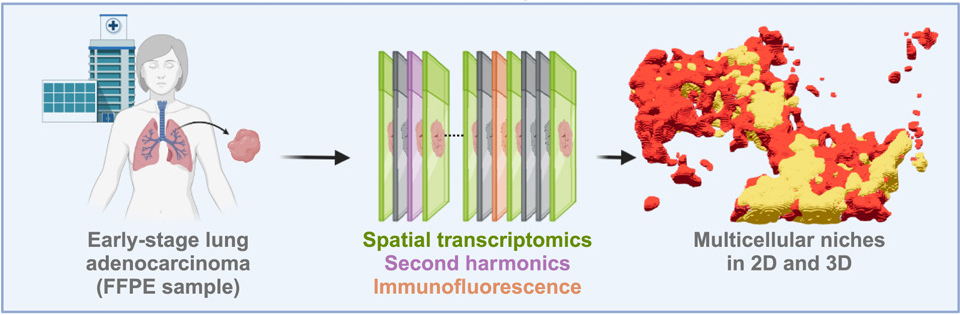
Für das Verständnis der Tumormikroumgebung sind nicht nur die vorhandenen Zellen entscheidend, sondern auch ihre genaue Umgebung und ihre Kommunikation untereinander. Ein internationales Forschungsteam um Nikolaus Rajewsky vom Berliner Institut für Medizinische Systembiologie des Max Delbrück Center (MDC-BIMSB) konnte das komplexe Zusammenspiel von Tumorzellen mit Hilfe räumlicher Transkriptomik nun nachvollziehen. Der methodische Ansatz ist in zwei Studien in Cell Systems beschrieben. Auch wenn die Analyse auf eine einzelne Gewebeprobe eines Lungentumors im Frühstadium beschränkt war, bietet die Machbarkeitsstudie hochpräzise Einblicke in die Tumorarchitektur und damit Angriffspunkte für eine personalisierte Krebstherapie.
Um das komplexe Ökosystem des Tumors zu erfassen, nahm das Forschungsteam zusammen mit dem Berliner Pathologie-Spezialisten Aignostics sowohl die Tumorzellen als auch die extrazelluläre Matrix in den Blick. Auf dieser Basis sollen sich anhand der präzisen und umfassenden Daten laut Rajewsky die molekularen Mechanismen des Phänotyps rechnerisch vorhersagen lassen: „Das ist wirklich neu – und von fundamentaler Bedeutung, wenn die personalisierte Medizin Wirklichkeit werden soll.“
Für die Analysen wurde aus 34 Schnitten einer FFPE-Gewebeprobe von nicht-kleinzelligem Lungenkarzinom (NSCLC) zunächst eine Karte der Zellen und ihrer RNA-Moleküle erstellt. 340.000 einzelne Zellen und 18 Zelltypen ließen sich auf diese Weise untersuchen. Gleich 960 verschiedene RNA-Moleküle konnten dank des CosMx Spatial Molecular Imagers gleichzeitig detektiert werden – eine Technologie der nun zu Bruker Nano Analytics gehörenden Firma NanoString. Ein neu entwickelter Algorithmus (STIM), der in einem Begleitartikel beschrieben ist und moderne Bildanalyseverfahren aus dem maschinellen Sehen auf biologische Daten anwendet, ermöglichte daraufhin die Rekonstruktion virtueller 3D-Gewebeblöcke. Mittels Second Harmonic Generation Imaging ließen sich schließlich auch die Hauptbestandteile der extrazellulären Matrix – Elastin und Kollagen – sichtbar machen und innerhalb der zellulären Mikroumgebung kartieren.
Auf diese Weise konnte die aktuelle Studie einen Gewebeumbau nachweisen, der mit mehr Kollagen und weniger Elastin einhergeht. Weitere Untersuchungen zeigten, welche molekularen Phänotypen von Fibroblasten daran beteiligt sind. Darüber hinaus gelang es Signalwege zu identifizieren, die in situ das Tumorwachstum verstärken und ein Eindringen von Immunzellen verhindern können. Auf Basis der Immunsuppression und Interaktion mit der extrazellulären Matrix konnte somit eine personalisierte Behandlung entwickelt werden – etwa mit Checkpoint- und Integrin-Inhibitoren.
Dank der Fülle an gewonnenen Informationen ist die nächste Generation der Pathologie nun erreicht – selbst jahrelang fixierten Proben lassen sich noch wichtige Erkenntnisse abgewinnen. Zukünftig sollen auch Proteine in die Analysen einfließen. Das Forschungsteam arbeitet momentan an weiteren 700 Tumorproben von 200 Personen, um den Ansatz weiterzuentwickeln.

 Molecular Partners AG
Molecular Partners AG Flinn.ai
Flinn.ai geralt/pixabay.com
geralt/pixabay.com