NEBNext ARTIC Kits: Zuverlässige SARS-CoV-2 Sequenzierung
So erhalten Sie in nur einem Tag und mit nur 30 min Hands-on-Zeit exzellente NGS-Libraries mit bester Genomsequenzabdeckung
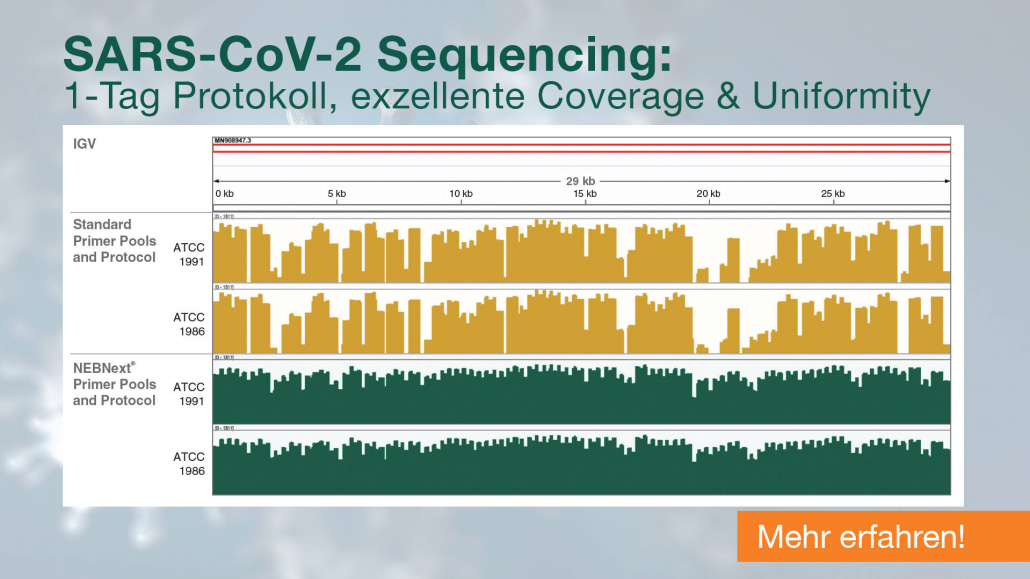
Mit Hilfe der neuen NEBNext® ARTIC Library Prep Kits präparieren Sie zuverlässig in nur einem Tag exzellente Libraries für die SARS-CoV-2 Sequenzierung auf Oxford Nanopore Technologies oder Illumina-Plattformen. Die Kits basieren auf dem ARTIC-Ansatz für Amplikon-Sequenzierung des kompletten viralen Genoms und wurden in Zusammenarbeit mit den Wissenschaftler*innen des ARTIC Netzwerkes (https://artic.network) entwickelt.
Der einzigartig ausbalancierte Primer-Pool gewährleistet eine verbesserte und gleichmäßige Abdeckung des Genoms – egal ob Sie 10 oder bis zu 10.000 Kopien des SARS-CoV-2 Genoms in die Reaktion einsetzen. Einfache und optimierte Protokollabläufe mit nur wenigen Pipettierschritten sowie spezielle Enzymformulierungen ermöglichen einen kurzen, ressourcenschonende Workflow bei konstanten RT- und PCR-Einstellungen. So entfallen zeit- und materialintensive RNA-Titrations- und Amplikon-Normalisierungsschritte (Illumina-kompatible Kits).
Mit den NEBNext ARTIC Kits erhalten Sie stets eine Library, die Ihnen exzellente Sequenzierdaten ohne sequenzspezifische „drop-outs“ ermöglicht.
Ihre Vorteile:
- Kompatibel – mit Illumina® oder Oxford Nanopore Technologies®
- Schnell – nur 30 min Hands-on-Zeit im 1-Tag Workflow
- Verlässlich – exzellente, gleichmäßige SARS-CoV-2 Genomabdeckung
- Effektiv – für variable Inputmengen von 10-10.000 Kopien
- Praktisch – gleiche RT Bedingungen für alle Input Mengen
- Einfach – Normalisierung der Amplikons vor der Library Prep entfällt (Illumina)
- Effizient – Kostengünstig & wirtschaftlich
Lesen Sie hier ein Statement unseres Early-Access-Users Dr. Vladimir Benes über die NEBNext ARTIC Kits:
“We have been impressed by the great performance of the NEB- Next ARTIC FS kit in our sequencing of human samples containing SARS-CoV-2 RNA. With this kit we have been successfully processing samples of varying quality and quantity and have been amazed by its sensitivity and remarkable uniformity of the viral genome coverage. The quick turnaround time of the robust workflow has been truly beneficial for us.”
Dr. Vladimir Benes, EMBL Heidelberg, Early-Access-User
Detaillierte Informationen finden Sie unter: www.neb-online.de/ARTIC


 MedtecLIVE
MedtecLIVE Athos Holding
Athos Holding BioSpring GmbH
BioSpring GmbH